Stimare correttamente i rapporti di parentela è fondamentale nel calcolo degli indici genetici e genomici e, soprattutto, per monitorare la consanguineità della popolazione. Due soggetti sono definiti “parenti” se hanno almeno un antenato in comune nel loro pedigree, mentre un soggetto è “consanguineo” se esiste una relazione di parentela tra i suoi genitori. Tradizionalmente, la parentela e la consanguineità erano stimate usando solo il pedigree, che ha il grosso vantaggio di poter essere stimato per tutti i soggetti iscritti al Libro Genealogico. Con il pedigree si stima la probabilità che due soggetti abbiano in comune, per discendenza, porzioni del loro patrimonio genetico. La stima della parentela da pedigree non è però in grado di identificare quali porzioni del genoma siano trasmesse da un soggetto ai suoi discendenti (nel caso di fratelli pieni la parentela da pedigree è di 0,5 e, quindi, condividono il 50% del loro genoma). L’avvento della genomica, invece, ci ha permesso di stimare la reale porzione di genoma condivisa tra i soggetti. Nel caso di fratelli pieni si osserva una distribuzione della parentela che varia da 0,2 (solo il 20% del genoma è in comune tra due fratelli pieni) a 0,7 (il 70% del genoma è uguale tra due fratelli pieni).
Il caso studio che vi presentiamo in questa breve sinossi è stato condotto su 3.581 giovani tori delle cinque razze da carne (Figura 1) selezionate da ANABIC (Marchigiana, Chianina, Romagnola, Maremmana e Podolica), in prova di performance dal 1985 al 2019. L’obiettivo del nostro lavoro è stato quello di valutare e confrontare il trend della consanguineità in 30 anni di selezione attraverso un approccio genetico (pedigree) ed un approccio genomico, che per una miglior comprensione chiameremo “new”.
Figura 1: le cinque razze bianche da carne italiane.
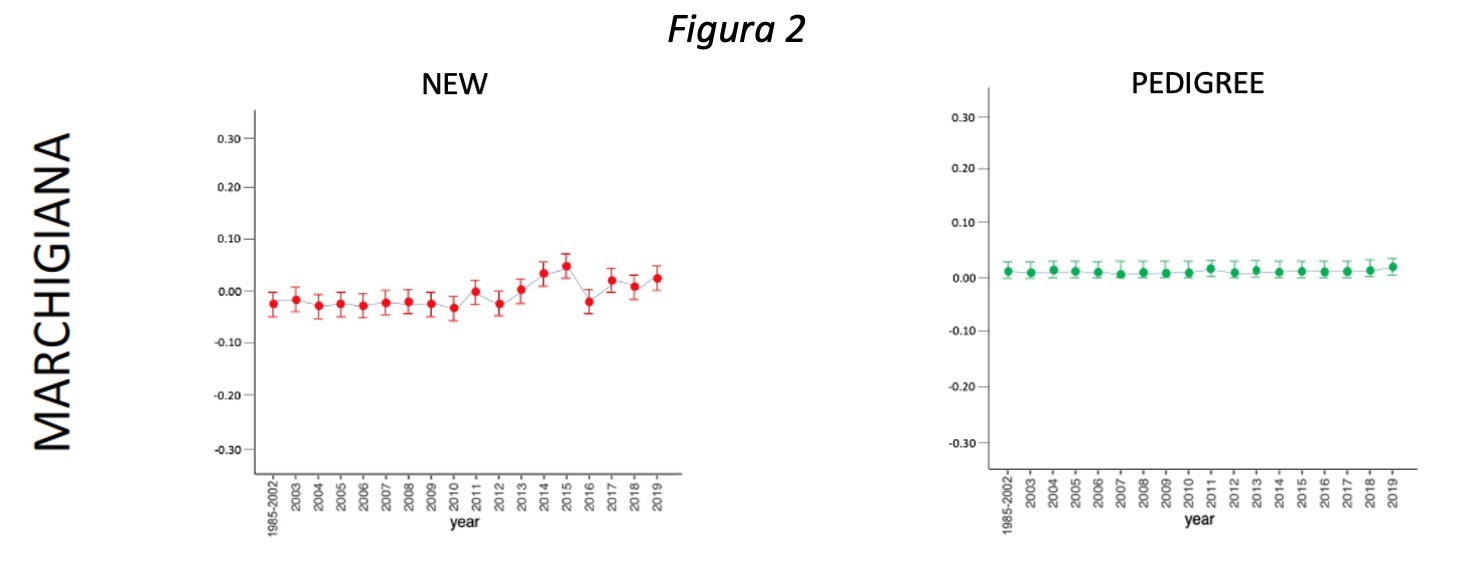
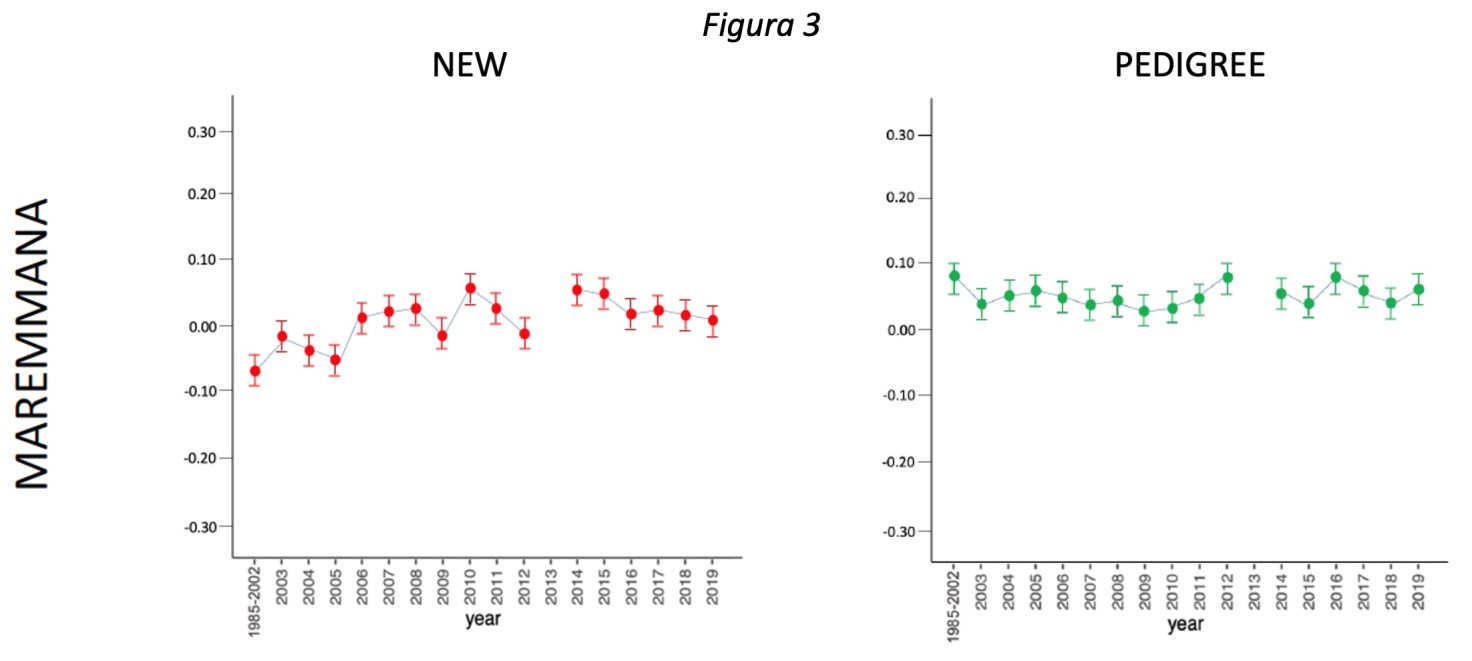
Abbiamo quindi calcolato, mediante programmi dedicati, il livello di consanguineità individuale e di popolazione per tutte le cinque razze oggetto di studio. La media dei valori annuali di popolazione è stata raffigurata graficamente per tutte le razze, ed è stata tracciata una linea tra i valori annuali che ci ha permesso di determinare l’andamento della consanguineità nelle 3 decadi in analisi. Riportiamo nelle figure 2 e 3, a titolo esemplificativo, l’andamento della consanguineità da pedigree e da genomica in una razza specializzata (Marchigiana) ed in una razza rustica (Maremmana). I valori medi complessivi di consanguineità sono stati anche riportati nella Tabella 1 per facilitarne la comprensione.
Tabella 1.
| RAZZA | PEDIGREE | NEW |
|---|---|---|
| MARCHIGIANA | 0.012 | 0.018 |
| CHIANINA | 0.018 | 0.024 |
| ROMAGNOLA | 0.023 | 0.027 |
| MAREMMANA | 0.054 | 0.062 |
| PODOLICA | 0.055 | 0.061 |
Come è intuibile dalle immagini e dalla tabella, i valori medi annui di ogni razza sono molto vicini allo zero e le oscillazioni nel corso degli anni sono minime. I valori più bassi sono stati rilevati nelle tre razze specializzate (0,012-0,027), ed in particolar modo nella razza Marchigiana (0,012-0,018). Nelle razze rustiche i valori di consanguineità sono risultati leggermente più elevati (0,054-0,062), ma comunque sempre molto vicini allo zero. Confrontando inoltre i valori ottenuti mediante pedigree e genoma, non abbiamo rilevato grandi differenze numeriche, anche questo a confermare l’ottima attività di selezione e programmazione degli accoppiamenti portata avanti da ANABIC negli anni.
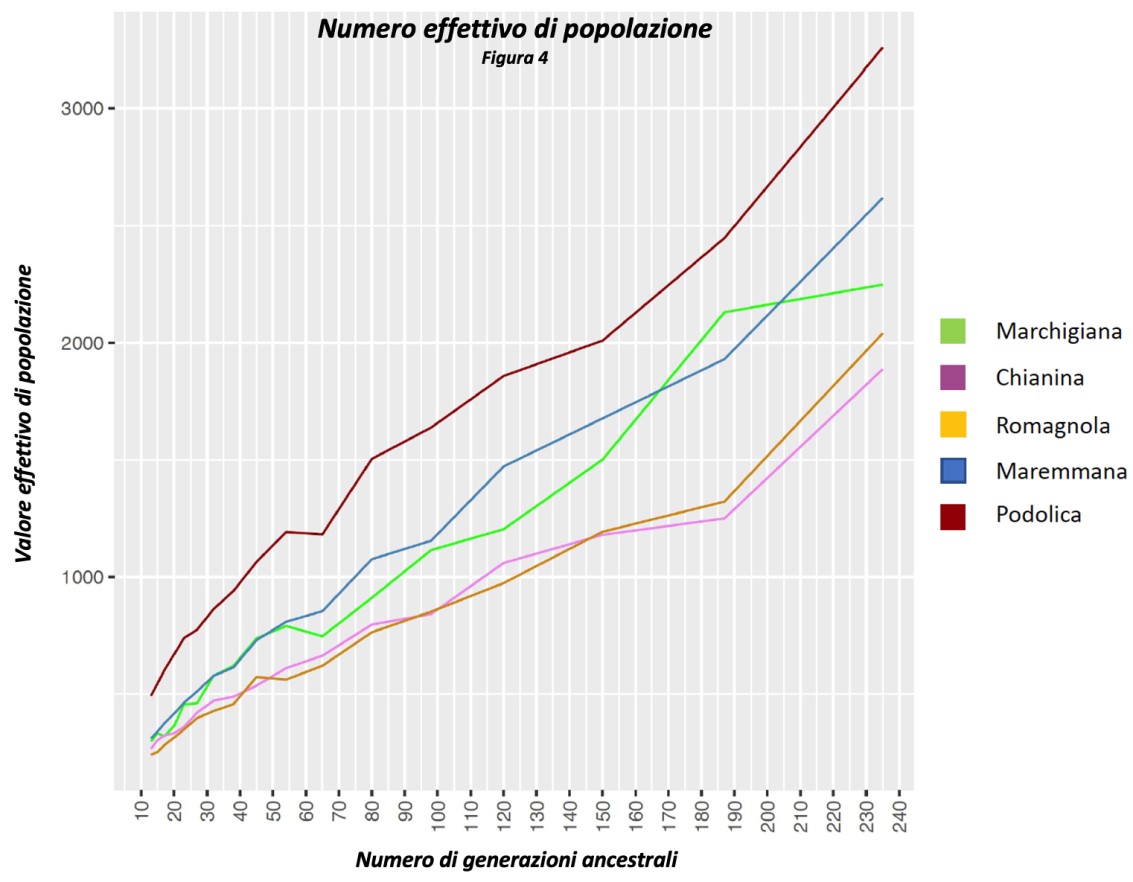
Successivamente, abbiamo calcolato i coefficienti di consanguineità individuali attraverso diversi metodi, che assegnano livelli di consanguineità differenti da zero ad individui con entrambi i genitori conosciuti. Tuttavia, se il numero di genitori sconosciuti è considerevole, i coefficienti di consanguineità individuali potrebbero essere gravemente sottostimati. Nel corso degli anni sono stati proposti diversi indicatori per riconoscere la diversità genetica delle popolazioni anche in presenza di alberi genealogici incompleti. Per esempio, è fondamentale tener sempre in considerazione i potenziali colli di bottiglia considerando il “numero effettivo di antenati” necessario per spiegare la completa diversità genetica della popolazione. La valutazione della dimensione effettiva di popolazione ha permesso di stimare la dimensione di una “popolazione ideale” che subisca gli effetti della consanguineità allo stesso grado della popolazione reale. Nelle cinque razze selezionate da ANABIC, abbiamo rilevato un calo marcato nella dimensione effettiva della popolazione. In linea di principio, questo calo marcato potrebbe essere causato dall’introduzione dell’inseminazione artificiale in cui un numero ridotto di tori si accoppia con un numero elevato di vacche. È importante però che questo declino non sia associato ad una riduzione dell’eterozigosità, che porterebbe ad avere più soggetti parenti nella popolazione. Nel caso studio qui presentato, l’eterozigosità è rimasta costante nel tempo, conseguenza anch’essa del fatto che le nostre popolazioni sono caratterizzate da animali poco parenti tra di loro.
Concludendo, possiamo affermare che il basso livello di consanguineità riscontrato in questo studio conferma il successo del programma di selezione dei bovini da carne italiani condotto da ANABIC, che mira da sempre a minimizzare la consanguineità, anche in considerazione dei suoi ben noti effetti negativi. Abbiamo osservato inoltre che l’incremento annuo di consanguineità nelle razze in analisi è inferiore a quanto riportato in altri studi simili al nostro condotti in diverse razze da latte degli Stati Uniti d’America e dell’Irlanda, probabilmente a causa di fattori legati alla gestione riproduttiva (alto numero di tori da riproduzione, accoppiamenti programmati per ridurre al minimo la consanguineità, ecc.). Essendo la prima volta che un approccio genomico viene applicato su giovani tori in performance al centro genetico ANABIC, non abbiamo evidenziato grandi differenze tra i valori di consanguineità da pedigree e da genomica. Siamo però sicuri del fatto che, con il passare del tempo, l’introduzione della genomica nei piani di selezione sarà di fondamentale importanza per effettuare una pre-selezione dei candidati in ingresso al centro genetico e, soprattutto, per garantire una graduale identificazione dei geni associati a salute, benessere, produttività e qualità dei prodotti, indispensabili per l’economia e la sostenibilità degli allevamenti.
Ulteriori approfondimenti relativi alle analisi qui descritte sono stati recentemente pubblicati nel nostro articolo sulla rivista “PeerJ – Life and Environment”: “Rovelli G, Luigi-Sierra MG, Guan D, Sbarra F, Quaglia A, Sarti FM, Amills M, Lasagna E. 2021. Evolution of inbreeding: a gaze into five Italian beef cattle breeds history. PeerJ 9:e12049. https://doi.org/10.7717/peerj.12049”.
Autori
Dott. Giacomo Rovelli e Prof. Emiliano Lasagna, Dipartimento di Scienze Agrarie, Alimentari e Ambientali – Università degli Studi di Perugia.