Un recente studio condotto da ricercatori dell’Università di Padova, in collaborazione con il Consiglio Nazionale delle Ricerche, ha dimostrato come utilizzando i dati provenienti dai controlli funzionali ed applicando metodi statistici avanzati sia possibile prevedere lo stato di salute della mammella bovina nei vari momenti della sua lattazione. Un esempio concreto di zootecnia di precisione focalizzato alla sostenibilità dell’allevamento delle bovine da latte.
La mastite, infiammazione della ghiandola mammaria in seguito ad infezione, costituisce uno dei problemi economici e sanitari più rilevanti negli allevamenti di bovine da latte a livello mondiale. La salute dell’apparato mammario delle bovine influenza la quantità e la qualità del latte prodotto, oltre che la sostenibilità dell’allevamento stesso. La salute della mammella viene valutata in modo preliminare attraverso la conta delle cellule somatiche (SCC, Somatic Cell Count) presenti nel latte, riconosciute quali indicatore indiretto di mastite [1]. Sebbene il controllo periodico delle SCC abbia consentito una riduzione delle infezioni negli allevamenti, la mastite non è stata del tutto eliminata. Si tratta di una patologia complessa, influenzata da molteplici fattori che, pertanto, richiede programmi di gestione sempre più innovativi.
Le SCC comprendono in realtà diversi tipi cellulari, ovvero i leucociti, che sono coinvolti nella risposta immunitaria dell’animale alle infezioni e includono linfociti, neutrofili e macrofagi, e le cellule epiteliali di sfaldamento. Conoscere la proporzione dei diversi tipi cellulari che costituiscono le SCC e che svolgono ruoli diversi durante l’infiammazione, può aiutare a definire in modo più accurato lo stato di salute della mammella. Mentre il latte proveniente da ghiandole mammarie sane contiene principalmente macrofagi e linfociti, i neutrofili costituiscono la popolazione cellulare predominante in presenza di infezione.
Di recente, è stato possibile implementare analisi avanzate del latte presso i laboratori dell’Associazione Regionale Allevatori del Veneto (ARAV), grazie all’acquisizione di un analizzatore innovativo ad elevate prestazioni per il conteggio delle cellule somatiche differenziali (DSCC, Differential Somatic Cell Count). Le DSCC rappresentano la proporzione combinata percentuale di neutrofili e linfociti, mentre la percentuale di macrofagi è pari a 100 – DSCC [2] e, in combinazione con le tradizionali SCC, possono fornire un quadro più dettagliato dell’effettivo stato infiammatorio della mammella [3].
L’accesso a una grande quantità di informazioni raccolte nell’ambito dei controlli funzionali mensili, comprendenti le SCC e le DSCC, offre la possibilità di applicare algoritmi di apprendimento automatico (machine learning) per l’analisi dei dati. Tali metodi consentono di identificare relazioni significative tra una variabile di risposta (es. stato di salute della mammella) ed una serie di altre variabili (es. SCC, DSCC, produzione di latte, ordine di parto, stadio di lattazione) e di sfruttare queste informazioni per “addestrare” diversi modelli di previsione e valutare le loro prestazioni predittive su dati sconosciuti. In sintesi, predire lo stato sanitario della mammella a partire da altre informazioni disponibili.
In un recente lavoro pubblicato nella rivista Scientific Reports, alcuni ricercatori dell’Università di Padova, in collaborazione con l’Istituto di Biologia e Biotecnologia Agraria del Consiglio Nazionale delle Ricerche, hanno confrontato otto diversi algoritmi di machine learning per prevedere lo stato di salute dell’animale al controllo funzionale successivo (SCC sopra o sotto 200.000 cellule/mL), a partire dai dati raccolti al controllo funzionale precedente (stadio di lattazione, ordine di parto, anno e stagione di campionamento, produzione e composizione del latte, SCC e DSCC, per un totale di 15 variabili). Il dataset iniziale, costituito da 18.442 controlli, è stato suddiviso in due set, uno utilizzato per le fasi di training e testing dei modelli (80% dei dati) e uno per la validazione esterna (il rimanente 20% dei dati). Nella fase di apprendimento (training) ciascun modello osserva, apprende le relazioni tra le variabili di input (i dati raccolti al controllo funzionale precedente) e l’output (la variabile binaria che vogliamo predire – SCC alte o basse al controllo successivo), e cerca di formulare delle previsioni sulla base di quanto imparato. Una volta terminata la fase di apprendimento, l’affidabilità previsionale degli algoritmi viene testata eseguendo la stessa procedura sul set di testing, per valutare l’eventuale presenza di sovradattamento (overfitting) con il set di training. Il set di testing ha la stessa struttura del set di training, ma dati diversi. L’uso di diversi set di testing ottenuti attraverso procedure di ricampionamento, consente una valutazione complessiva migliore della qualità di predizione. Una volta ottenute delle buone performances sui set di training e testing, è possibile testare ulteriormente i modelli su osservazioni nuove appartenenti al set di validazione esterna.
Tutti i metodi presi in esame hanno mostrato un’accuratezza superiore al 75% (Figura 1); ciò significa che in un allevamento di 100 animali è possibile prevedere correttamente lo stato di salute della mammella al controllo successivo di almeno 75 animali.
Figura 1. Confronto delle performances (accuratezza e valore di kappa) di otto diversi algoritmi machine learning [Linear Discriminant Analysis (LDA), Neural Network (NN), Generalized Linear Model (GLM), Support Vector Machines (SVM), Random Forest (RF), Classification and Regression Trees (CART), k-Nearest Neighbors (kNN) e Naïve Bayes (NB)] nel predire lo stato di salute della mammella nel set di testing.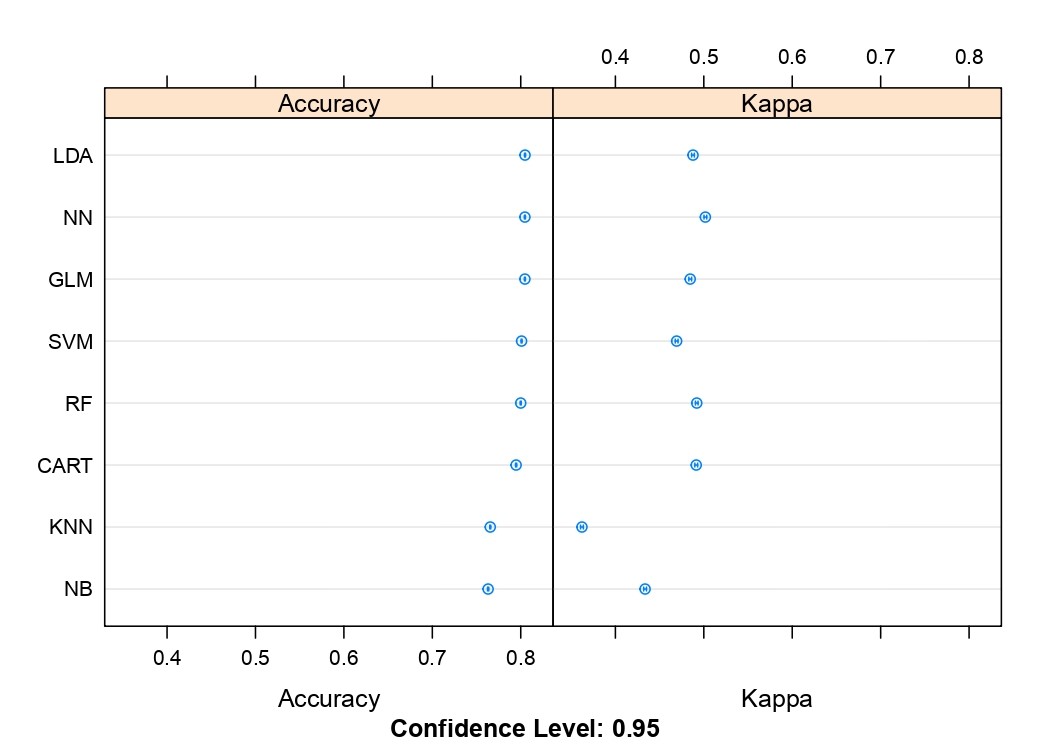
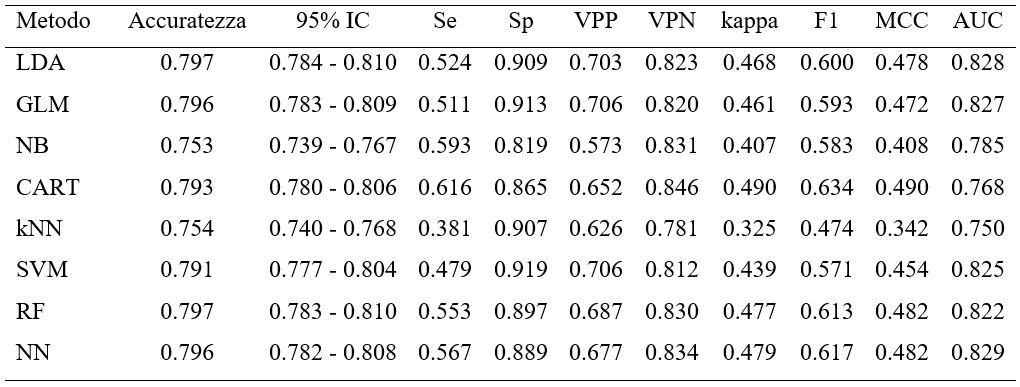
Tuttavia, tra tutti i metodi confrontati sulla base di diverse metriche, le reti neurali, il Random Forest ed i modelli lineari sono risultati i migliori nel predire lo stato di salute della mammella sia nel set di testing (Figura 1) che nel set di validazione esterna (Tabella 1).
Tabella 1. Metriche [accuratezza, intervallo di confidenza al 95% (95% IC), sensibilità (Se), specificità (Sp), valore predittivo positivo (VPP), valore predittivo negativo (VPN), valore di kappa, punteggio F1, coefficiente di correlazione di Matthew’s (MCC) e area sotto la curva (AUC)] per il confronto degli otto algoritmi machine learning applicati al set di validazione esterna per predire lo stato di salute della mammella. I modelli predittivi testati sono: Linear Discriminant Analysis (LDA), Generalized Linear Model (GLM), Naïve Bayes (NB), Classification and Regression Trees (CART), k-Nearest Neighbors (kNN), Support Vector Machines (SVM), Random Forest (RF) e Neural Network (NN).
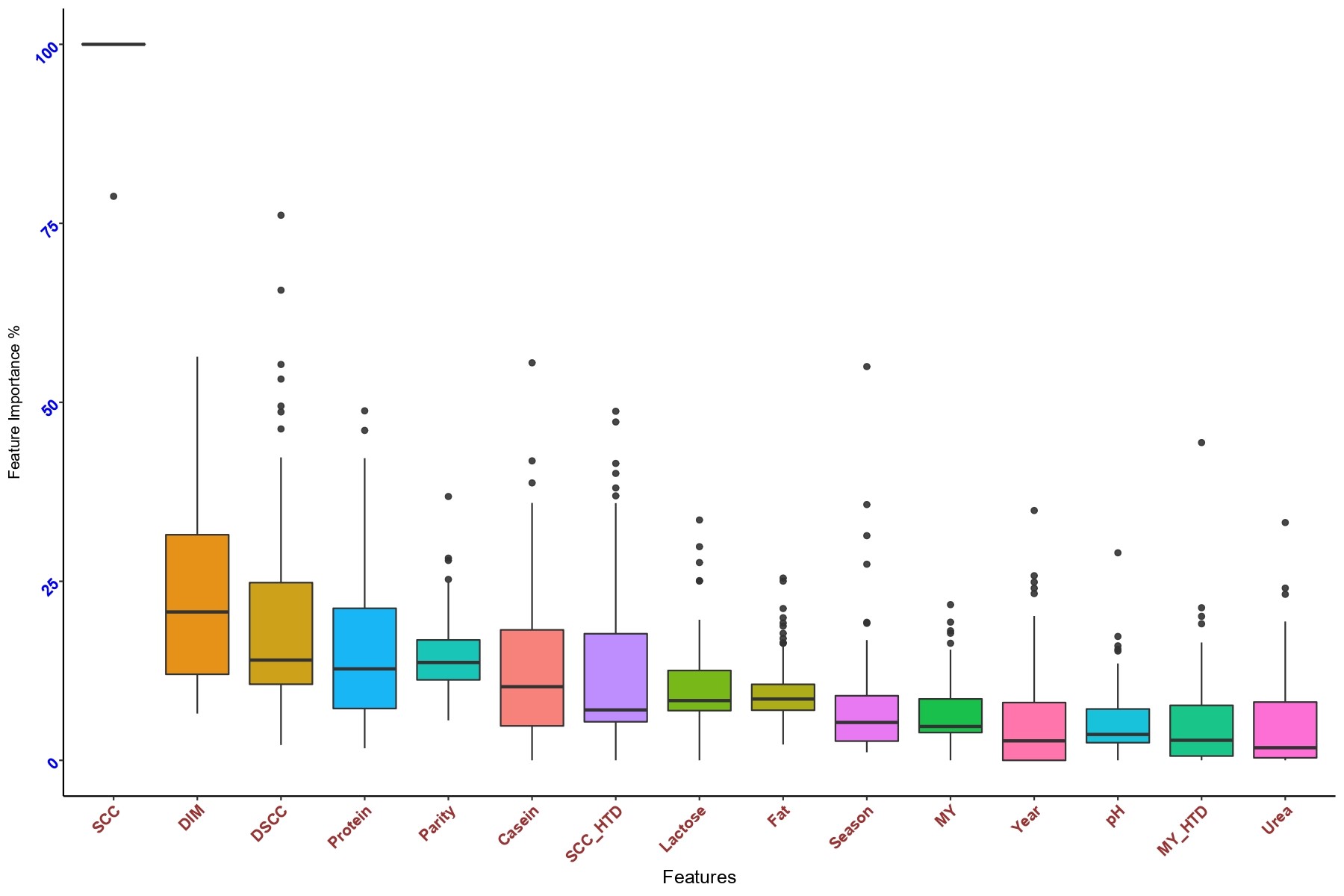
Inoltre, tra le 15 variabili prese in considerazione, le più significative nel predire lo stato di salute della mammella sono risultate essere, ovviamente, le SCC al controllo precedente, lo stadio di lattazione (noto fattore che influenza la variazione delle SCC) e le DSCC al controllo precedente (Figura 2).
Figura 2. Importanza delle variabili (espressa in percentuale, dove 100% rappresenta la variabile più significativa) nella predizione dello stato di salute della mammella nel set di testing attraverso l’uso delle reti neurali (NN). Le variabili considerate sono: conta delle cellule somatiche (SCC), stadio di lattazione (DIM), cellule somatiche differenziali (DSCC), proteina percentuale (Protein), ordine di parto (Parity), caseina percentuale (Casein), SCC medio del gruppo di bovine campionate lo stesso giorno nello stesso allevamento (SCC_HTD), lattosio percentuale (Lactose), grasso percentuale (Fat), stagione di campionamento (Season), produzione di latte (MY), anno di campionamento (Year), pH, produzione di latte media del gruppo di bovine campionate lo stesso giorno nello stesso allevamento (MY_HTD e urea).
Questi risultati confermano il valore aggiunto di conoscere la diversa proporzione dei tipi cellulari che compongono le SCC totali. Infatti, 149 animali su 384 che sono stati erroneamente classificati come sani, presentavano al controllo precedente SCC < 200.000 cellule/mL ma DSCC > 70%, a indicare presenza di infiammazione. Infatti, animali con basse SCC (per cui tradizionalmente verrebbero considerati sani) ma elevate DSCC presentano in realtà una risposta infiammatoria in atto e sono suscettibili alla mastite [4].
I risultati di questo studio suggeriscono come i metodi di machine learning siano promettenti per migliorare il processo decisionale in ambito zootecnico, aiutando l’allevatore nell’identificazione precoce di animali problematici e favorendo la riduzione dell’uso di antibiotici in stalla. Se le Associazioni Allevatori saranno interessate ad ottenere questa tipologia di informazione, sarà possibile sviluppare un tool applicativo.
Sinossi tratta da “Comparison of machine learning methods to predict udder health status based on somatic cell counts in dairy cows“, Scientific Reports 2021, 11, 13642, https://doi.org/10.1038/s41598-021-93056-4.
Bibliografia
[1] Harmon, R. J. Somatic cell counts: a primer. Proc. Natl. Mastitis Coun. 40th Annual Meeting, Feb 11–14, 2001 Reno, NV, pp 3–9 (2001).
[2] Damm, M., Holm, C., Blaabjerg, M., Bro, M. N. & Schwarz, D. Differential somatic cell count – a novel method for routine mastitis screening in the frame of Dairy Herd Improvement testing programs. J. Dairy Sci. 100(6), 4926–4940 (2017).
[3] Bobbo, T., Penasa, M. & Cassandro, M. Combining total and differential somatic cell count to better assess the association of udder health status with milk yield, composition and coagulation properties in cattle. Ital. J. Anim. Sci. 19, 697–703 (2020).
[4] Schwarz, D. et al. Microscopic differential cell counts in milk for the evaluation of inflammatory reactions in clinically healthy and subclinically infected bovine mammary glands. J. Dairy Res. 78(4), 448–455 (2011).
Autori
Tania Bobbo1,*, Stefano Biffani2, Cristian Taccioli3, Mauro Penasa1 & Martino Cassandro1,4
1Dipartimento di Agronomia, Animali, Alimenti, Risorse naturali e Ambiente, Università degli Studi di Padova, Legnaro (PD), Italia
2Istituto di Biologia e Biotecnologia Agraria, Consiglio Nazionale delle Ricerche, Milano, Italia
3Dipartimento di Medicina Animale, Produzioni e Salute, Università degli Studi di Padova, Legnaro (PD), Italia
4Associazione Nazionale Allevatori della Razza Frisona Bruna e Jersey Italiana, Cremona, Italia
*Nuova affiliazione: Dipartimento di Scienze Agrarie e Ambientali – Produzione, Territorio, Agroenergia, Università degli Studi di Milano, Milano, Italia